-
微小RNA(microRNA, miR)是一种短链非编码RNA,能特异性结合靶基因mRNA的3′非编码区域,从转录水平或转录后水平调控基因表达。人体细胞中miR-374a编码基因位于人染色体Xq13.2区域。其在细胞和组织中的异常表达与肿瘤的发生有重要关系。在肺腺癌细胞中,miR-374a的过表达,能使靶基因TGFA的表达下调,从而抑制细胞的增殖、转移和侵袭[1]。在非小细胞肺癌中,能通过靶向结合LARP1,减弱LARP1启动子效应,下调STAT3信号通路,从而抑制细胞增殖、转移[2]。但在乳腺癌细胞中miR-374a能抑制Wnt/β-catenin信号通路负调控因子,促进癌细胞转移。在骨肉瘤和食管癌组织中能够抑制靶基因的表达,促进细胞的增殖和转移,同样具有原癌基因作用[3-4]。此外,miR-374a的异常表达与炎症性肠病有关,对保护细胞的化学性缺氧损伤也有作用[5-6]。miR-374a分子表达在细胞中发挥着广泛作用,有许多功能有待深入研究。本文就miR-374a低表达细胞系的构建及鉴定作一探讨。
-
克隆载体pMD18T为TaKaRa公司产品;pSpCas9(BB)-2A-Puro(PX459)购于Addgene公司。质粒提取试剂盒、胶回收试剂盒购于AXYGEN公司;miRNA及Total RNA提取试剂盒为TIANGEN公司的产品;逆转录酶使用的是TaKaRa公司的Reverse Transcriptase M-MLV(RNase Hˉ);所用引物均由上海生工生物工程有限公司合成;细胞系HEK-293T,A549,感受态细胞Stable由本实验室留存提供。定量PCR仪为Eppendof公司的Realplex。
-
选择基因组miR-374a成熟体附近200 bp的序列为目标序列,通过使用CRISPR设计工具(http://tools.genome-engineering.org)在线设计靶标序列,选择评分较高更靠近miR-374a成熟体位点的sgRNA序列。针对sgRNA序列(见表 1)合成引物。退火后用T4连接酶与经BbsⅠ酶切的pSpCas9(BB)-2A-Puro载体连接,16 ℃,30 min。将50 μL冰置融化的Stable感受态细胞加入连接体系中,冰置10 min,42 ℃,90 s热休克处理后,冰浴2 min,经复苏后铺在含有氨苄青霉素的LB培养基上37 ℃恒温过夜培养,挑选单克隆扩大培养,提取质粒,交由上海生工生物工程有限公司进行序列测定[7]。
名称 序列 gRNA primer-F CAC CTA AGT GCT ATA ACA CTT ATC gRNA primer-R AAA CGA TAA GTG TTA TAG CAC TTA 374a-genome-primer-F GGT AGT AGG GTG GGG AGG TTA AGA GG 374a-genome-primer-R GAC TGA AAT CCA AAC ACA AAA ATG GC miR-374a stem-loop RT Primer GTC GTA TCC AGT GCA GGG TCC GAG GTA TTC GC ACT GGA TAC GAC CAC TTA TCA G miR-374a forward Primer CGA TCG CGC TTA TAA TAC AAC C U6 stem-loop Primer GTC GTA TCC AGT GCA GGG TCC GAG GTA TTC GCA CTG GAT ACG ACA AAA ATA TG U6 forward Primer CGC AAA TTA GTG AAG CGT TC stem-loop RT universal Primer GTG CAG GGT CCG AGG TAT TC TGFA-qPCRP1 CCT GTT CGC TCT GGG TAT TGT TGFA-qPCRP2 CAG ATT CCC ACA CTC AGT TCT GC GAPDH-qPCRP1 TGG GTG TGA ACC ATG AGA AGT ATG GAPDH-qPCRP2 ACT GTG GTC ATG AGT CCT TCC A 表 1 所用的引物和序列
-
人胚肾HEK-293T细胞及肺癌细胞系A549细胞培养于1640培养基(含10%胎牛血清、1%青霉素-链霉素、2 mmol/L谷氨酰胺),参照说明书。其中A549细胞转染质粒48 h后加入嘌呤霉素(10 μg/mL)加压筛选。继续培养48 h,待细胞大面积死亡后,更换新鲜培养基,继续培养。所得细胞进行传代,用于细胞冻存和后续的检测。
-
胰酶消化并收集上述转染入基因编辑载体的HEK-293T细胞,用裂解液(0.5% SDS、0.1 mol/L NaCl、0.05 mol/L EDTA、0.01 mol/L pH 8.0 Tris-Cl)和蛋白酶K。50 ℃裂解3 h。加入200 μL 6 mol/L饱和NaCl溶液,冰浴10 min后离心。取上清液后,加入等体积酚-三氯甲烷抽提,离心后取上清液,向上清液中加入800 μL无水乙醇和1/10体积的3M乙酸钠,颠倒混匀后离心。弃掉上清液后用800 μL 70%预冷乙醇洗涤。待干燥后,用适量水溶解沉淀基因组。
-
根据在基因组上的靶位点,在基因组序列上下游300 bp左右,设计鉴定引物(序列见表 1),用PCR Taq Mix(博迈德),扩增,反应条件为94 ℃ 3 min(94 ℃ 30 s 55 ℃ 30 s 72 ℃ 45 s)×30 cycle 72 ℃ 5 min。PCR产物电泳,回收(AxyPrep DNA Gel Extraction Kit)约600 bp产物与pMD18T连接,转化入感受态DH5α细菌。随机挑取10个克隆,摇菌,提取质粒,送至公司进行序列测定。
-
MiR-374a相对表达含量检测方法参考课题组前期使用的miRNA定量方法文献:以未经转染的A549细胞作为对照细胞,经过Cas9-miR374a质粒转染和加压筛选的细胞为实验组,每组均做3个重复。使用TIANGEN公司试剂盒提取两组细胞中的miRNA,用Nanodrop-1000进行RNA定量,用U6和miR-374a茎环结构引物(序列见表 1),进行逆转录,以U6做内参,用SYBR Green Realtime PCR Master Mix(Toyobo)进行实时定量PCR,采用2-ΔΔCt法计算上述2组细胞的miR-374a相对表达量。
-
根据文献报道,TGFA为miR-374的靶基因。通过IGC(http://biodb.swu.edu.cn/qprimerdb/)在线设计合成TGFA引物(序列见表 1),收集细胞提取总RNA,用oligdT为引物,M-MLV逆转录,逆转录方法参照说明书,以得到的cDNA为模板,GAPDH为内参,qPCR方法检测A549细胞和Cas9-miR374a细胞中TGFA的相对表达量,采用2-ΔΔCt法进行分析。
-
以未经转染的A549为对照组,经miR374a转染、加压筛选细胞系为实验组细胞。监测不同时间点(24 h、48 h、72 h)细胞生长状况,绘制生长曲线。具体方法如下:收集上述2组细胞,用Invitrogen公司的Countess对细胞自动计数。以每孔5 000个/孔的细胞数量铺至96孔板。在检测的不同时间点,提前3 h加入CCK8(10 μL DoJinDo)。继续培养3 h,取出,使用BioTek公司的多功能酶标仪,在λ=450 nm检测细胞吸光度值(每组5个重复)。比较研究不同时间点细胞的增殖活力。
-
采用t检验。
-
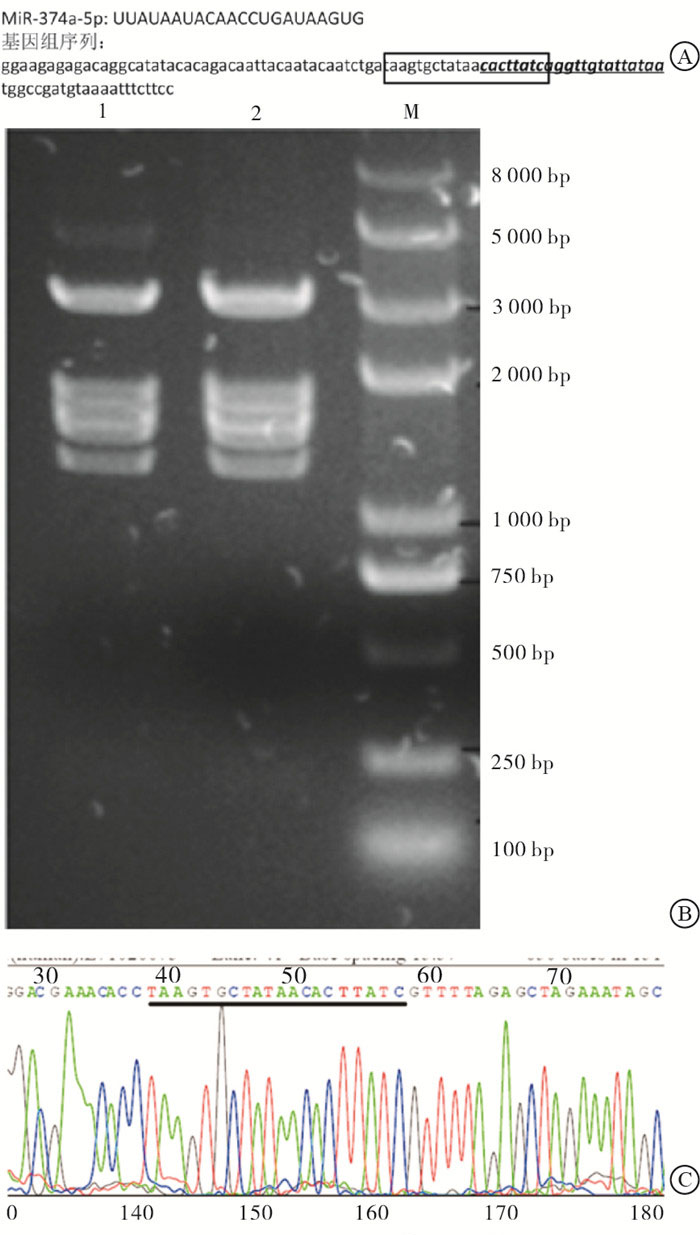
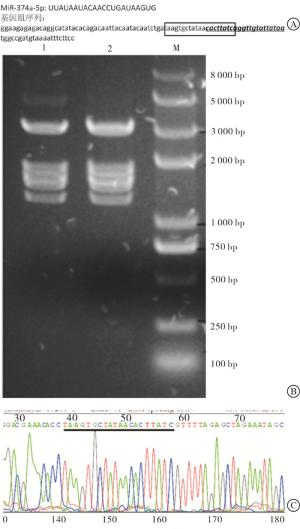
在线设计靶标序列,选择评分较高、更靠近miR-374a成熟体位点的sgRNA序列(见图 1A)。将sgRNA引物退火回收后与BbsⅠ酶切后pSpCas9(BB)-2A-Puro载体连接,紫外光下分析电泳结果,酶切条带与理论一致(见图 1B)。将鉴定正确的1#或2#质粒交由公司测序,结果显示目标序列插入了基因编辑载体中(见图 1C)。表明miR374a基因编辑载体成功构建。
-
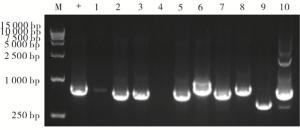
为了检测miR-374a基因编辑载体对细胞基因组水平的影响,我们将上述成功构建的miR-374a基因编辑载体转染入HEK-293T细胞中。48 h收集的细胞,提取基因组。以提取的基因组为模板,用PCR方法对含基因编辑位点的基因组序列进行扩增。胶回收扩增产物后,与pMD-18T载体相连。随机挑选10个单克隆菌,进行PCR菌落鉴定(见图 2)。结果扩增出大小不一的条带,分析可能为细胞的基因组发生不同类型插入或缺失突变的基因编辑的结果而至。将上述克隆提取的质粒交由公司进行序列测定。测序结果和目标基因组序列进行比对。结果提示:转染基因编辑载体的细胞中目标位点10个克隆有9个发生了缺失或插入突变。提示:针对miR-374a成熟体区域的基因编辑载体可以对该区域的基因组进行有效的识别和编辑。
基因编辑的细胞基因组扩增产物连接于pMD-18T后,随机选择10个单克隆,进行PCR鉴定的结果(图中+为对照细胞基因组扩增的产物,M为Trans15K的DNA分子量标准,1~10为10个克隆扩增产物)。
-
上述结果提示我们找到了针对miR-374a成熟体区域有效的靶位点。为了进一步检测基因编辑后细胞miR-374a的表达情况。我们将上述载体转染入目标细胞系人肺癌细胞系A549细胞中,经过嘌呤霉素的加压筛选,扩增存活细胞后收集细胞。以未转染细胞为对照细胞,提取收集到的细胞miRNA。定量检测基因编辑组细胞miR-374a成熟体的相对表达,结果提示转染基因编辑载体的细胞miR-374a表达量明显降低(P < 0.01)(见表 2)。因此针对miR-374a成熟体附近位点基因组的基因编辑可以有效地降低miR-374a成熟体的表达水平。
分组 n Hsa-miR-374a相对表达量 t P control
Cas9/crispr-374a3
31.001 471±0.053 974
0.313 673±0.109 2779.77 < 0.01 表 2 基因编辑对细胞中miR-374a表达含量的影响
-
选择TGFA为检测目标,设计针对该分子的定量引物。以未转染细胞为对照,提取上述转染细胞的总RNA。Olig dT为引物进行逆转录后,通过qPCR检测2组细胞TGFA相对表达。以GAPDH为内参进行相对表达量校正。结果提示,miR-374a基因编辑载体转染组TGFA的表达量明显升高(P < 0.05)(见表 3)。提示构建miR-374a低表达细胞对靶基因的TGFA表达抑制作用减弱,结果符合预期。进一步说明了我们建立起了miR-374a低表达细胞。
分组 n TGFA mRNA相对表达量 t P control
Cas9/crispr-374a3
31.001 95±0.063 201
5.901 12±2.068 9744.10 < 0.05 表 3 经过miR-374a基因编辑的A549细胞TGFA靶基因表达变化(x±s)
-
对比检测上述筛选的细胞与未转染细胞的细胞活力,结果在A549细胞中基因编辑载体转染的细胞增殖能力明显高于对照细胞(P < 0.05~P < 0.01)(见表 4)。而在人胚肾细胞HEK-293T细胞中,转染的细胞则比对照细胞增殖活力明显下降(P < 0.01)(见表 5)。说明对细胞基因组miR-374a的基因编辑引起其表达下降影响到了细胞的增殖活性。
分组 24 h 48 h 72 h Control 0.95±0.02 1.53±0.05 3.20±0.26 Cas9/crispr-374a 1.22±0.04 2.29±0.04 3.66±0.05 t 10.78 17.92 2.96 P < 0.01 < 0.01 < 0.05 表 4 MiR-374a表达下降对A549细胞增殖的影响(x±s)
分组 24 h 48 h 72 h Control 0.550 6±0.007 54 0.938 6±0.022 2 1.944±0.040 8 Cas9/crispr-374a 0.455 8±0.006 22 0.621 4±0.009 4 1.173±0.017 3 t 16.79 22.78 30.13 P < 0.01 < 0.01 < 0.01 表 5 MiR-374a表达下降对HEK-293T细胞增殖的影响(x±s)
-
MiR-374a目前被证明在肿瘤发生和转移、炎症性肠病及缺氧损伤中具有重要作用。前期研究发现在病原感染细胞中miR-374a受到了异常调控,但其功能及在病原致病中的作用有待深入研究。miRNA功能丧失是研究miRNA分子功能较为有效的实验方法之一。基于CRISPR/Cas9的基因编辑方法,是一种依赖于特定RNA序列的编辑系统,通过具有靶标作用的gRNA序列与Cas9编码基因共表达实现特定位点的基因修饰,可以实现基因敲除或敲入。其中Cas9蛋白对特定位点的DNA切割能引发易错修复,导致细胞基因组水平的突变。gRNA是一种能够指导crRNAs和反式RNAs结合的独立小分子,导致靶基因的3′端三联体密码子NGG即PAM序列上游特定位点的DNA断裂,从而导致同源介导的修复。基于RNA指导的CRISPR/Cas9系统能对不同基因组进行高效编辑,更加省时省力,具有更大的发展前景和实际用途[8]。本研究正是基于该技术对miRNA基因组序列进行靶向编辑,以期通过破坏其DNA分子序列,降低miRNA分子的有效转录表达。
在本次实验中,为了研究miR-374a在细胞中的功能,构建miR-374a低表达A549细胞系,我们选择了与miR-374a成熟体基因序列部分重叠的基因片段作为靶标。设计合成gRNA相关序列构建入基因编辑载体pSpCas9(BB)-2A-Puro中。该载体转染细胞后可实现sgRNA与Cas9蛋白的共表达。成功鉴定载体后,将该载体分别转染入HEK-293和A549细胞,经过嘌呤霉素的加压筛选,获得可能被基因编辑的细胞。进而在基因组水平分析了细胞中miR-374a的基因组序列。并对细胞中miRNA及其靶基因表达水平进行了检测。结果提示含有靶标序列的CRISPR/Cas9系统,成功实现了miR-374a成熟体基因组区域的基因编辑。在靶向位点附近发生了多种类型的基因插入和缺失突变。对细胞中miR-374a相对表达检测结果提示, miR-374a表达含量明显降低,分析可能是由于插入或缺失突变造成了编码miR-374a基因区域突变,并且前体颈环结构的破坏而不能形成成熟体的缘故。以miR-374a靶基因TGFA作为检测目标,检测其在细胞中的相对表达,结果提示发生基因编辑的细胞miR-374a靶基因TGFA表达增加,与预期一致。在细胞活性实验中,我们对人肺癌细胞系A549和人胚肾HEK-293T细胞进行检测,发现miR-374a对不同的细胞增殖活力影响不同,分析可能是由于不同的细胞系各自的基因表达谱不一致,致使miR-374a在作用靶基因时具有不用的倾向性的原因。但其分子的作用和具体机制还待进一步深入研究。
上述研究结果说明,我们使用CRISPR/Cas9技术构建了miR-374a基因低表达细胞系,为进一步研究该分子的功能奠定了基础。
基于CRISPR/Cas9技术的miR-374a低表达细胞系的构建及鉴定
Constructing and identifying of cell line with low-expression miR-374a based on CRISPR/Cas9 system
-
摘要:
目的运用CRISPR/Cas9技术构建miR-374a低表达细胞系。 方法设计针对miR-374a成熟体gRNA,合成DNA,插入pSpCas9(BB)-2A-Puro,将鉴定正确的载体转染入肺癌细胞系A549细胞,使用嘌呤霉素筛选转染的细胞;PCR方法扩增,序列测定分析细胞基因组中靶位点区域序列;定量PCR方法检测转染细胞中miR-374a及其靶基因TGFA的表达情况;细胞增殖实验检测miR-374a基因编辑对细胞生长情况的影响。 结果构建了靶向miR-374a成熟体基因序列的基因编辑载体,转染细胞经过加压筛选,序列分析结果提示靶向位点的基因发生了缺失或插入突变;定量PCR结果显示,转染基因编辑载体的细胞miR-374a表达水平较对照细胞明显下降(P < 0.01),而靶基因TGFA表达则明显上升(P < 0.05);细胞增殖实验显示miR-374a低表达的细胞增殖能力发生了明显改变(P < 0.05~P < 0.01)。 结论成功构建了miR-374a低表达细胞系,为后续研究该分子的功能提供了基础。 -
关键词:
- 微小RNA-374a /
- 转化生长因子 /
- CRISPR/CAS9技术 /
- 细胞增殖
Abstract:ObjectiveTo establish the cell line with low-expression miR-374a using CRISPR/CAS9 gene targeting technology. MethodsThe mature gRNA targeting miR-374a was designed to synthesize the DNA, which was inserted into the CRISPR/CAS9 vector to construct the pSpCas9(BB)-2A-Puro.A549 cells were transfected with the constructed vector, and screened using puromycin.The gene sequences of target site were analyzed using PCR and sequencing method.The qPCR was used to detect the relative expression levels of miR-374a and its target gene TGFA in transfected cells.The CCK8 was used to detect the proliferation activity of cells. ResultsThe gene targeting miR-374a vector was successfully constructed.The transfected cells were screened under pressure, and the sequencing results showed that the inserting/deletion mutation was found.The results qPCR showed that the expression level of miR-374a significantly decreased, and the expression level of TGFA significantly increased in transfecting gene editing vector compared with control cells(P < 0.01 and P < 0.05).The cell proliferation assay showed that the proliferation activity significantly changed in low-expressed miR-374a cells(P < 0.05 to P < 0.01). ConclusionsThe low-expressed miR-374a cell line was successfully established, which provides the basis in studying the molecular function. -
Key words:
- microRNA-374a /
- transforming growth factor alpha /
- CRISPR/CAS9 /
- cell proliferation
-
表 1 所用的引物和序列
名称 序列 gRNA primer-F CAC CTA AGT GCT ATA ACA CTT ATC gRNA primer-R AAA CGA TAA GTG TTA TAG CAC TTA 374a-genome-primer-F GGT AGT AGG GTG GGG AGG TTA AGA GG 374a-genome-primer-R GAC TGA AAT CCA AAC ACA AAA ATG GC miR-374a stem-loop RT Primer GTC GTA TCC AGT GCA GGG TCC GAG GTA TTC GC ACT GGA TAC GAC CAC TTA TCA G miR-374a forward Primer CGA TCG CGC TTA TAA TAC AAC C U6 stem-loop Primer GTC GTA TCC AGT GCA GGG TCC GAG GTA TTC GCA CTG GAT ACG ACA AAA ATA TG U6 forward Primer CGC AAA TTA GTG AAG CGT TC stem-loop RT universal Primer GTG CAG GGT CCG AGG TAT TC TGFA-qPCRP1 CCT GTT CGC TCT GGG TAT TGT TGFA-qPCRP2 CAG ATT CCC ACA CTC AGT TCT GC GAPDH-qPCRP1 TGG GTG TGA ACC ATG AGA AGT ATG GAPDH-qPCRP2 ACT GTG GTC ATG AGT CCT TCC A 表 2 基因编辑对细胞中miR-374a表达含量的影响
分组 n Hsa-miR-374a相对表达量 t P control
Cas9/crispr-374a3
31.001 471±0.053 974
0.313 673±0.109 2779.77 < 0.01 表 3 经过miR-374a基因编辑的A549细胞TGFA靶基因表达变化(x±s)
分组 n TGFA mRNA相对表达量 t P control
Cas9/crispr-374a3
31.001 95±0.063 201
5.901 12±2.068 9744.10 < 0.05 表 4 MiR-374a表达下降对A549细胞增殖的影响(x±s)
分组 24 h 48 h 72 h Control 0.95±0.02 1.53±0.05 3.20±0.26 Cas9/crispr-374a 1.22±0.04 2.29±0.04 3.66±0.05 t 10.78 17.92 2.96 P < 0.01 < 0.01 < 0.05 表 5 MiR-374a表达下降对HEK-293T细胞增殖的影响(x±s)
分组 24 h 48 h 72 h Control 0.550 6±0.007 54 0.938 6±0.022 2 1.944±0.040 8 Cas9/crispr-374a 0.455 8±0.006 22 0.621 4±0.009 4 1.173±0.017 3 t 16.79 22.78 30.13 P < 0.01 < 0.01 < 0.01 -
[1] WU H, LIU Y, SHU XO, et al.MiR-374a suppresses lung adenocarcinoma cell proliferation and invasion by targeting TGFA gene expression[J].Carcinogenesis, 2016, 37(6):567. doi: 10.1093/carcin/bgw038 [2] XU Z, XU Z, XU J, et al.LARP1 is regulated by the XIST/miR-374a axis and functions as an oncogene in non-small cell lung carcinoma[J].Oncol Rep, 2017, 38(6):3659. [3] LU T, ZHANG C, CHAI MX, et al.MiR-374a promotes the proliferation of osteosarcoma cell proliferation by targeting Axin2[J].Int J Clin Exp Pathol, 2015, 8(9):10776. [4] WANG Y, XIN H, HAN Z, et al.MicroRNA-374a promotes esophageal cancer cell proliferation via Axin2 suppression[J].Oncol Rep, 2015, 34(4):1988. doi: 10.3892/or.2015.4182 [5] XIONG Y, QIU J, LI C, et al.Fortunelin-induced modulation of phosphatase and tensin homolog by microRNA-374a decreases inflammation and maintains intestinal barrier function in colitis[J].Front Immunol, 2018, 9(83):1. [6] GONG W, QIE S, HUANG P, et al.Protective effect of mir-374a on chemical hypoxia-induced damage of PC12 cells in vitro via the GADD45α/JNK signaling pathway[J].Neurochem Res, 2017, 43(3):581. [7] RAN FA, HSE PD, WRIGHT J, et al.Genome engineering using the CRISPR-Cas9 system[J].Nat Protoc, 2013, 8(11):2281. doi: 10.1038/nprot.2013.143 [8] GAJ T, GERSBACH CA, BARBAS CF 3rd.ZFN, TALEN and CRISPR/Cas-based methods for genome engineering[J].Trends Biotechnol, 2013, 31(7):397. doi: 10.1016/j.tibtech.2013.04.004 -




 下载:
下载: